微軟研究院近期宣布了一項突破性進展,推出了一款名為BioEmu-1的人工智能模型,該模型能夠預測蛋白質隨時間變化的運動和形狀演變,為生物醫學、藥物發現以及結構生物學領域帶來了全新的研究視角。
蛋白質,這一生命的基本構建塊,在生物體的各種過程中扮演著不可或缺的角色,從肌肉的形成到疾病的防御,無處不在。近年來,科學家利用深度學習技術,在解析蛋白質結構方面取得了顯著成就,能夠基于氨基酸序列準確預測蛋白質的三維結構。然而,這種預測往往局限于靜態結構,就像電影中的一幀畫面,無法展現蛋白質的動態特性。
與DeepMind的AlphaFold專注于靜態結構預測不同,BioEmu-1致力于模擬蛋白質在不同構象之間的動態轉換。這一特性使得BioEmu-1成為理解蛋白質運動機制、設計高效治療方案的重要工具。雖然AlphaFold 3在結構生物學領域取得了重要進展,改進了蛋白質與其他分子的相互作用模型,但它仍然無法預測蛋白質隨時間的動態變化。
BioEmu-1通過生成式深度學習技術,從大型數據集中學習蛋白質結構的模式,并生成與這些模式相符的新樣本。它的訓練結合了靜態蛋白質結構數據、分子動力學模擬數據以及實驗穩定性數據,從而能夠更全面地理解蛋白質的動態行為。BioEmu-1的核心機制是一個擴散模型,它通過迭代生成蛋白質結構,并根據學習到的約束條件不斷提高預測的準確性。
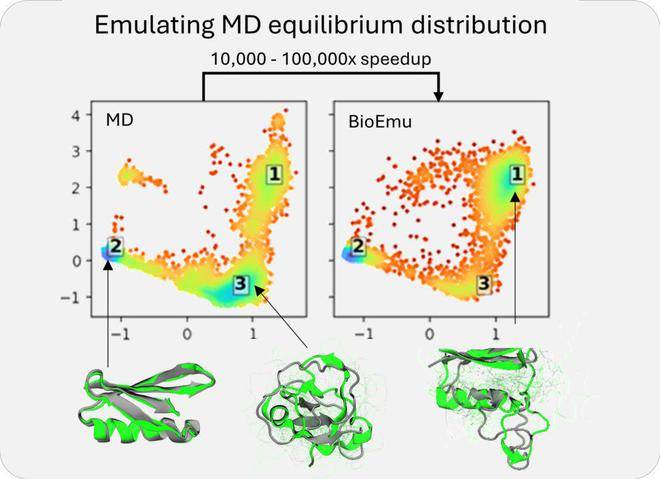
BioEmu-1的輸出包括平衡系綜的預測和自由能的預測,這些預測對于理解蛋白質的穩定性和功能至關重要。為了訓練BioEmu-1,微軟研究院使用了三種類型的數據集:AlphaFold數據庫的結構數據、廣泛的分子動力學模擬數據集以及實驗性蛋白質折疊穩定性數據集。通過這些數據集的訓練,BioEmu-1能夠識別蛋白質序列與多個不同結構之間的映射關系,預測合理的結構變化,并以正確的概率對折疊和未折疊結構進行采樣。
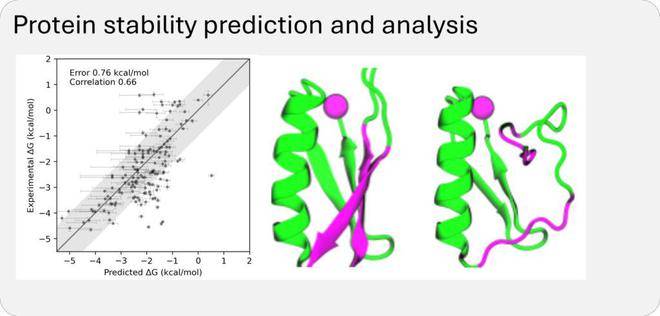
BioEmu-1的高效性也是其一大亮點。它每小時能夠生成數千個蛋白質結構樣本,這一速度遠遠超過了傳統分子動力學模擬的數周時間。同時,BioEmu-1預測自由能的誤差幅度在1 kcal/mol以內,與傳統分子動力學模擬相當,但計算成本卻顯著降低。這意味著研究人員可以更快、更經濟地探索蛋白質的動態行為,加速新藥的開發進程。
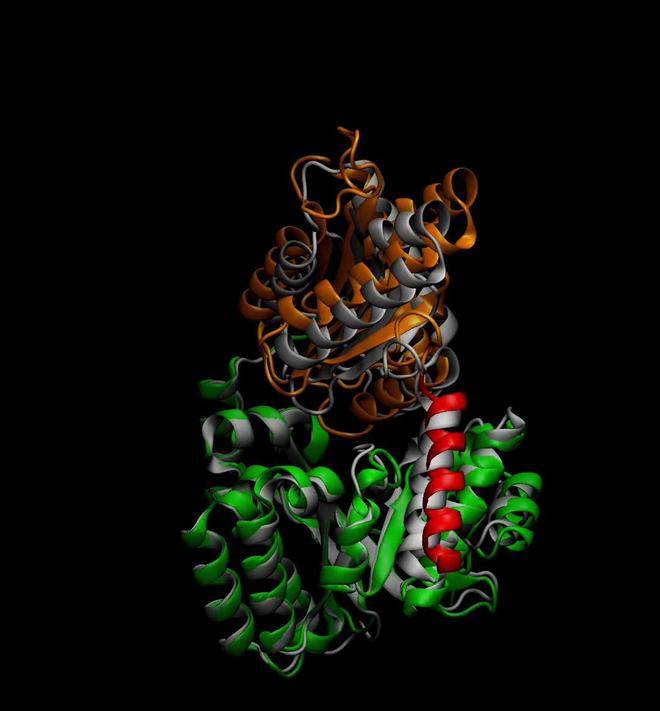
微軟研究院的這一創新成果,無疑為蛋白質科學研究開辟了新的道路。BioEmu-1的推出,不僅提高了我們對蛋白質動態行為的理解,還為藥物發現和生物醫學研究提供了強有力的支持。隨著BioEmu-1的廣泛應用,我們有理由相信,未來在蛋白質相關領域的研究將取得更加顯著的進展。






